fMRI brain image preprocessing(SPM12)
June 20, 2023
Convert, FieldMap , 以及Preproceesing
Convert DICOM - NIfTI #
两种方法: SPM12 DICOM import 和 MRIcroGL(dcm2niix)
直接使用 SPM12 转换非常简单,但缺点是无法看到 DICOM 文件的原始数据(比如TR,TE),可能会对 Fieldmap 校正选择参数带来一些困难。而使用 MRIcroGL 则可以额外生成保留原数据的 json 文件。因此这里主要说明 MRIcroGL 的操作方法。
MRIcroGL is also used for visualizing imaging data, especially group-level results. 但目前只用来转换成 NIfTI 文件.
以下内容参考了 Andysbrainbook.
安装 MRIcroGL #
安装后进入软件,菜单栏选中import->Convert DICOM to NIfTI,会看到如下界面:
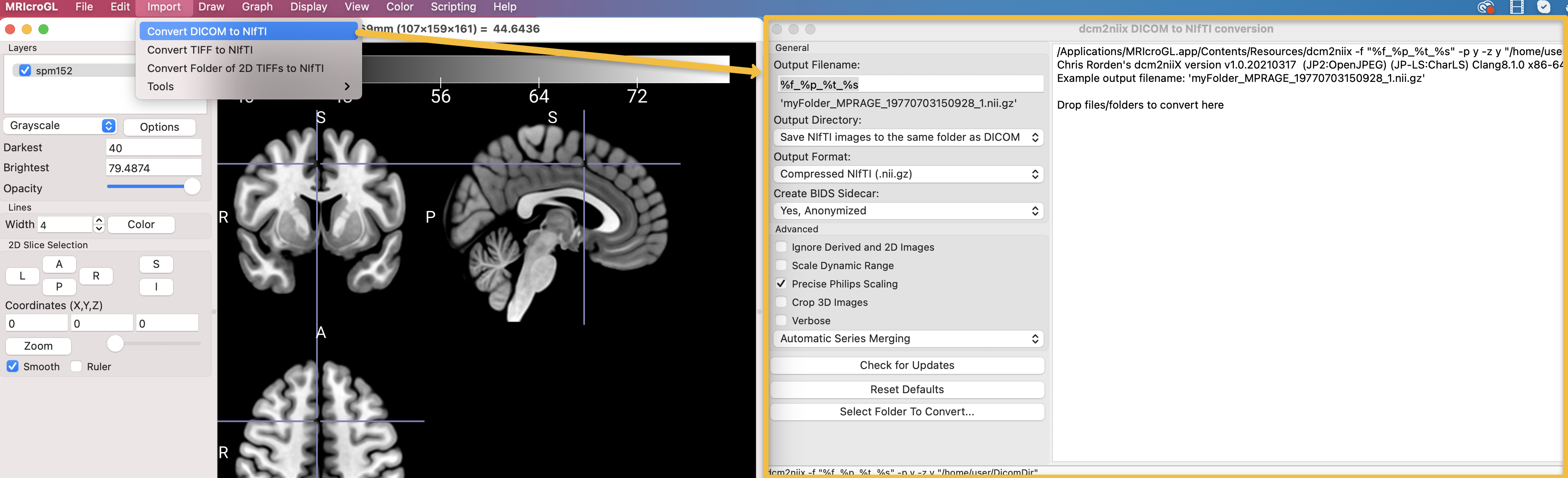
Output Filename:默认是格式化运算符%f_%p_%t_%s,鼠标停留在名称上会有每个变量的具体说明,可以根据需要修改转换后输出的名称。
Output Derictiory:输出路径,默认与DICOM文件在同一个文件夹。
Output Format:输出格式,默认是compressed NIfTI. 但请注意,SPM无法打开压缩的文件(AFNI和FSL都可以打开),建议手动选择uncompressed NIFTI。
Create BIDS Sidecar:该选项会创建一个包含原DICOM数据的Javascript Object Notation (.json)文件,包括slice-timing, slice order, the repetition time, echo time 等等。其中,Echo time 是 Fieldmap 时很重要的一个参数。
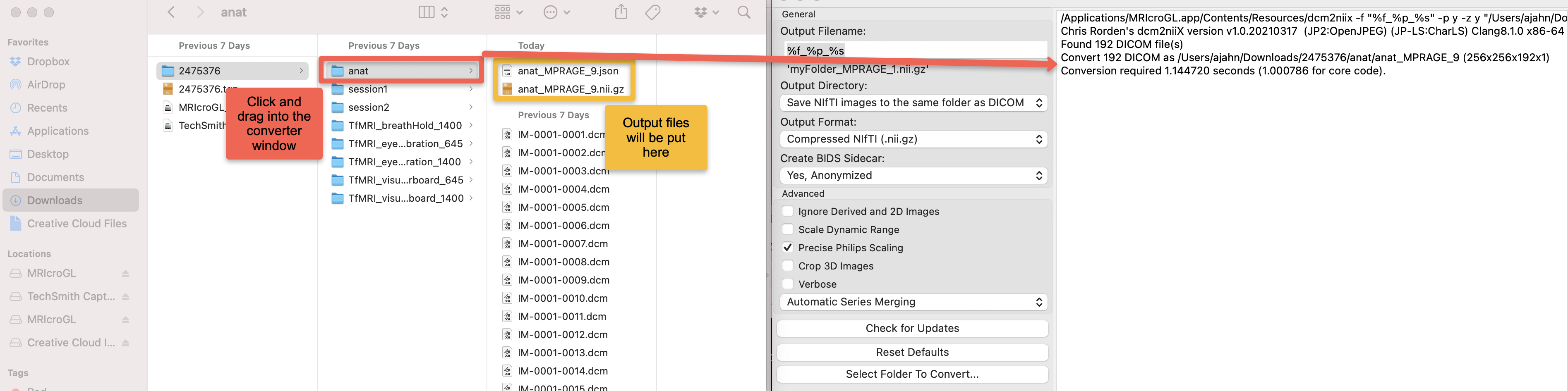
Converting the DICOM Files using MRIcroGL #
可以通过鼠标拖拽或者Select Folder to Convert 按钮选择想要转换的DICOM文件夹,右侧窗口会显示转换完成。
Running DICOM to NIFTI from the Command Line #
(还没使用过)
FieldMap 校正 (在一切开始之前) #
FieldMap 校正是指,对成像进行动态或静态的畸变校正。
SPM12 中使用 Fieldmap toolbox 进行 Fieldmap 校正。
两个步骤:
- Calculate,计算 VDM(voxel displacement map)
- Apply,使用 VDM 对成像进行校正
几个我用到的关键参数:
- Calculate VDM.
Magnitude Image: 有多个时选择 TE 更小的,有更好的信号强度。
Phase Image:信号和radiofrequency pulse的相位。
Echo time:需要手动输入 Magnitude Image 和 Phase Image 的 short TE 和 long TE。(i.e.,[5.16 7.62])
Mask brain: 选择是否 mask brain。如果选择 masking,Magnitude Image 会用来生成一个 brain mask。
Blip direction:相位编码方向与 Y 方向(从后脑勺往前额)的关系。+ve 为1,-ve 为 -1。
Total EPI readout time: 69.299
EPI-based filed map: 是否基于 EPI 获得的 Fieldmap,基于 EPI 的fieldmap 相当于是在扭曲的空间里获得了场图,因此生成 VDM 将会被调整。(non-EPI)
uflags: phase unwrapping 和 field map 处理的不同选项。加权平滑。
- Unwrapping method: Mark3D
- FWHM: 10
- Weighted smoothing: Weighted smoothing
mflags:segmentation 和创建 brain mask 的不同选项。使用 T1 对变形的 EPI 进行校正。
- Template images: toolbox/FieldMap/T1.nii
- FWHM: 5
- Number of erosions: 2
- Number of dilations: 4
- Threshold: 0.5
- Regularization: 0.02
EPI Sessions
- Select EPI to Unwarp: EPI第一张
- Match VDM to EPI: match VDM
VDM filename extension:生成的 VDM 文件的前缀,session。
Write unwarped EPI?: 导出校正后的 EPI,前缀是 u。(仅用来quality control of correction) [None]
计算 VDM 需要 MI 和 PI 这两个参数
- Apply VDM.
images:需要进行场图校正的 EPI。【代码写法】
Field map (vdm*file):需要应用的VDM,一个以vdm开头的文件。
P.S. 所有过程都可以以 batch code 方式批量操作。
校正后的EPI文件的前缀是wu,表示"warped and unwarped"。
校正后的EPI文件保存在与输入EPI图像相同的路径下。
Realignment (EPI) #
运动校正,修正EPI的位移
Realign面板里有四个选项:
- Realign (Estimate)
- Realign (Reslice)
- Realign (Est & Res)
- Realign & unwarp
其中 Realign & unwarp 主要负责修正fMRI scanner里的扭曲,现在的 MRI scanner 可以生成很完美的cubes,因此已经不常用了。
Estimate:计算为了匹配序列里第一张图片每张图片的移动和旋转参数。(只改变 image 的 metadata 和 headder info)
Reslice: 根据 Estimate 生成一系列新的 realigned images。(改变 image)
如果只是在 individual brain 层面进行 realign ➡️ smooth ➡️ do stats:进行 Realign (Est & Res)。 如果要 warp image to normalized space,在 normalized space 进行 realign:只进行 Realign (Estimate)。
Realign & Unwarp(运动校正和磁场畸变校正)
Unwarp 和 Field map
都是用于磁场畸变校正的方法,但具有不同的原理和实施步骤,具体根据实验的需要选用。
Unwarp(畸变校正):Unwarp方法是基于图像处理的技术,通过图像重采样和插值来校正图像畸变。它依赖于相邻时间点的fMRI图像信息。Unwarp方法通常需要额外的参考数据(例如,额外的fMRI序列)来估计磁场畸变的模型。
Field Map(场图法):Field map方法是基于磁场测量的技术,通过测量磁场变化来估计和校正磁场畸变。它依赖于额外的磁场映射扫描数据。
Data - Session
每个session内保持 Data / Data + phase map 一致
Images:所有EPI
Phase map (VDM * file)
Estimation Options
Filename prefix:u
【EPI第一张被修改】
Coregistration #
将T1登陆到EPI(变换、计算处理) 有三种变换方式:Affine(平移+旋转的刚体变换),Stretching(保持一个方向不变移动其他方向),Shearing(扩大+缩小变形)
-
Estimate:estimate用于估计将源图像(EPI)与参考图像(T1)对齐所需的变换参数。这些变换参数描述了源图像与参考图像之间的平移(translation)和旋转(rotation)关系,以使它们在相同的坐标空间中对齐。estimate步骤不会对图像进行重采样。
-
Reslice:reslice用于根据先前估计的变换参数对源图像 EPI 进行重采样,以使其与参考图像 T1 在空间上对齐。通过重采样,源图像 EPI 的像素将根据变换参数重新分布和插值,使其与参考图像 T1 的空间坐标一致。在后续分析时,EPI 与 T1 就可以在相同的空间坐标系下进行对应。
-
Estimate & Reslice:estimate & reslice 是将上述两个步骤合并为一个步骤的选项。一次性完成 estimate 和 reslice。
一些我用到的参数:
Reference Image: 其他图像配准时的对照. Batch 操作时一般选择 Realign: Estimate & Resllice: Mean image,即上一步Realign过后生成的 Mean image。(我的分析中跳过了Realignment,直接用了 T1 )
Source Image: 应基于参比图像进行处理的图像。我的分析中使用了EPI(f 开头)
Estimation options 中的参数:
-
Cost Function: Coregistration 涉及到最大化或最小化目标函数的参数(更好的Coregistation)常用的目标函数有:Mutual Information(mi),Normalized Mutual Information(nmi),Sum of Squared Differences(ssd),Correlation Ratio(corr_ratio). (我的分析用了mni) -
Separation: The average distance between sampled points (mm). 也就是源图像和参考图像之间的初始平移和旋转变换的初始步长。较大的Separation值适用于粗略的初始对齐,较小的Separation值适用于较精细的初始对齐。可以是一个vector,允许粗略的coregistration过后进行精细的coregsitration。i.e.,[4,2] -
Tolerances: 指控制配准算法的终止条件。这些参数指定了在搜索对齐参数空间时用于终止搜索的阈值或容差值。包括Translation Tolerances(平移容差), Rotation Tolerances(旋转容差), Scale Tolerances(缩放容差), Affine Tolerances(仿射容差). 通常情况下,建议开始时使用较大的容差值进行初始对齐,然后逐渐减小容差值以提高对齐的精度。 -
FWHM: FWHM参数定义了高斯滤波器的半高全宽(mm),用于指定 fMRI 图像的平滑程度。较小的FWHM值表示较小的滤波器,产生较少的平滑效果;较大的FWHM值表示较大的滤波器,产生更显著的平滑效果。较小的FWHM值适用于高分辨率数据和对细节较敏感的分析,但可能会保留更多的噪声。较大的FWHM值适用于低分辨率数据和对噪声较不敏感的分析,但可能会损失一些图像细节。i.e.,[7,7]
【EPI第一张被修改】
Segmentation #
将T1分解为标准脑6要素,分割后称为 TPM
相关的参数:
Channel: 指每个体素(Voxel)在fMRI数据中的观测通道数。通常为1,表示每个体素只有一个观测通道(T1)。
-
Volumes: 从已经选择的channel中选择scan。(这里就选择T1 images) -
Bias regularisation: 用于对磁共振图像中的偏差进行正则化的参数。偏差通常是由磁场非均匀性或硬件特性引起的亮度变化。通过启用偏差正则化,可以减少这些偏差的影响,提高图像质量。 MR扫描中的intensity variation可能由两种可能引起:MR扫描物理原理引起的偏差伪影(bias artifact),或者不同组织的特性。[light regularisation: 0.001] -
Bias FWHM: 指定了在偏差校正过程中应用的滤波器的全宽半最大值(Full Width at Half Maximum,FWHM)。较高的FWHM值表示较大的平滑程度,而较低的FWHM值表示较小的平滑程度。该参数控制偏差场的平滑程度。[60 mm cutoff] -
wirte:channel.write = [0 1]表示保存白质和灰质通道的分割结果。(Channel 0:gray matter, Channel 1:white matter, Channel 2:cerebrospinal fluid.)
tissue
Tissue probability map:
Segmentation生成文件在T1文件夹里,c1+T1文件名.nii
Normalisation #
EPI标准化
为了把 Coregistration 过后的图像和 MNI 标准脑 TPM 重合而进行计算,让所有图像都在一个 template 上,从而可以直观地 voxel by voxel 比较。
Smoothing #
EPI空间平滑化
通过应用高斯滤波器,对fMRI图像进行空间平滑处理。平滑处理有助于减少噪声和局部变异,提高信号与噪声的比值,并提升数据的统计可靠性。
空间上幅的表示方法:半值全幅(FWHM,Full Width at Half Maximum)
半值全幅(FWHM),指从 peak 开始高度一半地方的宽度. FWHM的值一般是voxel的2-3倍,不过在评价杏仁核这样的小结构时,也用1.5倍这样的小数值. FWHM越大,整体的功能画像就会越模糊(因为FWHM越大,意味着图像和平均的相邻voxel相关度变高).
我自己用到的顺序 #
Day1:
-
Convert DICOM to NIfTI
-
Fieldmap process (by BatchCalVDM.m)
-
Realign & Unwarp (including Apply VDM. By Batch_Realign_VDM.m)
-
Coregister EPI to T1 (by BatchCoreg.m) (EPI是filedmap校正过的)
-
Segmentation(x)
-
Smooth 555
Day2
-
Convert DICOM to NIfTI
-
Coregister EPI to T1 (by BatchCoreg.m)
-
Fieldmap process (by BatchCalVDM.m)
-
Coregister fieldmap-corrected EPI to T1
-
Segmentation
-
Normalisation
-
Smooth
Normalisation is used to compare d1 and d2 data.
Model construction
Estimation
construct contrasts
average among subjects (only D2)